ОБОБЩЕННЫЕ КРИТЕРИИ ИДЕНТИФИКАЦИИ ХИМИЧЕСКИХ СОЕДИНЕНИЙ МЕТОДАМИ ХРОМАТОГРАФИИ – МАСС-СПЕКТРОМЕТРИИ
Аннотация
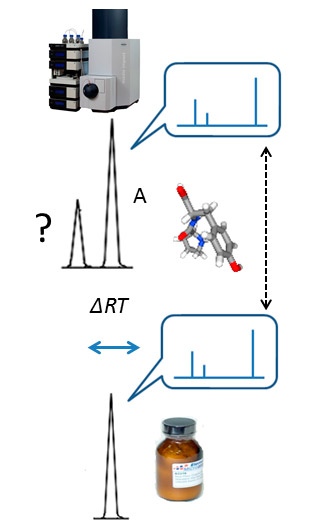
Представлен обзор критериев химической идентификации, применяемых в рамках целевого и нецелевого анализа методами хроматографии – масс-спектрометрии. Окончательные критерии в обоих случаях сопоставимы и предполагают близкое сходство характеристик хроматографического удерживания (времена или относительные времена) и масс-спектров (массы отдельных ионов, относительные интенсивности их пиков) идентифицируемых аналитов и аналитических стандартов. Сходство, как правило, означает максимально допустимые отклонения значений указанных величин, установленных для аналитов, от соответствующих справочных значений, приписанных известным соединениям. Используемые критерии идентификации включены в общие руководства по химическому анализу, подготовленные международными или национальными регулирующими организациями. В случае нецелевой идентификации аналиты и соответствующие стандарты неизвестны заранее. На первых этапах решения задач этого вида необходимо отобрать соединения − кандидаты на идентификацию, затем сравнить характеристики аналитов и соответствующих аналитических стандартов, используя критерии идентификации, аналогичные тем, которые сформулированы для целевого анализа. Для поиска кандидатов используют хроматографию и тандемную масс-спектрометрию высокого разрешения, продвинутые методы компьютерной обработки и предсказания данных, поиски в химических базах данных, сводках индексов удерживания и масс-спектральных библиотеках. Применение этих методов и процедур – условие надежности поиска кандидатов на идентификацию. Прогресс в области нецелевого анализа зависит от усовершенствования масс-спектрометрической техники, в первую очередь за счет повышения точности и воспроизводимости определения масс ионов. Не меньшее значение имеет развитие компьютерных технологий (информатики), что должно обеспечить появление более полных библиотек масс-спектров и более точных методов предсказания масс-спектрометрических и хроматографических данных.
Ключевые слова: химическая идентификация, масс-спектрометрия, хроматография, компьютеры, количественные критерии идентификации
Полный текст:
PDF (Russian)Литература
REFERENCES
Milman B.L., Zhurkovich I.K. Big data in modern chemical analysis. J. Anal. Chem., 2020, vol. 75, no. 3, pp. 443-452. DOI: org/10.1134/S1061934820020124.
Milman B.L. Vvedenie v khimicheskuiu identifikatsiiu [Introduction to Chemical Identification]. SPb: VVM, 2008. 180 p. (in Russian).
Milman B. L. Chemical identification and its quality assurance. Berlin: Springer, 2011. 281 p.
Watson D.G. A rough guide to metabolite identification using high resolution liquid chromatography mass spectrometry in metabolomic profiling in metazoans. Comput. Struct. Biotechnol. J., 2013, vol. 4, no. 5, p. e201301005. Available at: https://doi.org/10.5936/csbj.201301005 (Accessed 20 May, 2020).
Smith R., Mathis A.D., Ventura D., Prince J.T. Proteomics, lipidomics, metabolomics: a mass spectrometry tutorial from a computer scientist's point of view. BMC Bioinf., 2014, vol. 15, no. 7, p. S9. Available at: https://doi.org/10.1186/1471-2105-15-S7-S9 (Accessed 20 May, 2020).
Milman B.L. General principles of identification by mass spectrometry. TrAC Trends Anal. Chem., 2015, vol. 69, pp. 24-33. DOI: org/10.1016/j.trac.2014.12.009.
De Vijlder T., Valkenborg D., Lemière F., Romijn E.P., Laukens K., Cuyckens F. A tutorial in small molecule identification via electrospray ionization ‐ mass spectrometry: The practical art of structural elucidation. Mass Spectrom. Rev., 2018, vol. 37, no. 5, pp. 607-629. DOI: org/10.1002/mas.21551.
Godzien J., De la Fuente A.G., Otero A., Barbas C. Metabolite Annotation and Identification. Compr. Anal. Chem., 2018, vol. 82, pp. 415-445. DOI: org/10.1016/bs.coac.2018.07.004.
Molyneux R.J., Beck J.J., Colegate S.M., Edgar J., Gaffield W., Gilbert J., Hofmann T., McConnell L.L., Schieberle P. Guidelines for unequivocal structural identification of compounds with biological activity of significance in food chemistry (IUPAC Technical Report). Pure Appl. Chem., 2019, vol. 91, no. 8, pp. 1417-1437. DOI: org/10.1515/pac-2017-1204.
Zubarev R.A., Makarov A. Orbitrap mass spectrometry. Anal. Chem., 2013, vol. 85, no. 11, pp. 5288-5296. DOI: org/10.1021/ac4001223.
Gruber B., Weggler B.A., Jaramillo R., Murrell K.A., Piotrowski P.K., Dorman F.L. Comprehensive two-dimensional gas chromatography in forensic science: A critical review of recent trends. TrAC Trends Anal. Chem., 2018, vol. 105, pp. 292-301. DOI: org/10.1016/j.trac.2018.05.017.
Nováková L., Svoboda P., Pavlík J. Ultra-high performance liquid chromatography. Liquid Chromatography. Fundamentals and Instrumentation. Elsevier, 2017, pp. 719-769. DOI: org/10.1016/B978-0-12-805393-5.00029-4.
Tang D.Q., Zou L., Yin X.X., Ong C.N. HILIC‐MS for metabolomics: An attractive and complementary approach to RPLC‐MS. Mass Spectrom. Rev., 2016, vol. 35, no. 5, pp. 574-600. DOI: org/10.1002/mas.21445.
Blaženović I., Kind T., Ji J., Fiehn O. Software tools and approaches for compound identification of LC-MS/MS data in metabolomics. Metabolites, 2018, vol. 8, no. 2, p. 31. Available at: https://doi.org/10.3390/metabo8020031 (Accessed 21 May, 2020).
Matyushin D.D., Sholokhova A.Y., Karnaeva A.E., Buryak A. K. Various aspects of retention index usage for GC-MS library search: A statistical investigation using a diverse data set. Chemom. Intell. Lab. Syst., 2020, vol. 202, p. 104042. Available at: https://doi.org/10.1016/j.chemolab.2020.104042 (Accessed 21 May, 2020).
Milman B.L., Ostrovidova E.V., Zhurkovich I.K. Isomer differentiation using in silico MS2 spectra. A case study for the CFM-ID mass spectrum predictor. Mass Spectrom. Lett., 2019, vol. 10, no. 3, pp. 93-101. DOI: 10.5478/MSL.2019.10.3.93.
Milman B.L., Zhurkovich I.K. The chemical space for non-target analysis. TrAC Trends Anal. Chem., 2017, vol. 97, pp. 179-187. DOI: org/10.1016/j.trac.2017.09.013.
Milman B.L., Zhurkovich I.K. Mass spectral libraries: A statistical review of the visible use. TrAC Trends Anal. Chem., 2016, vol. 80. pp. 636-640. DOI: org/10.1016/j.trac.2016.04.024.
Analytical quality control and method validation procedures for pesticide residues analysis in food and feed (2019). Available at: https://ec.europa.eu/food/sites/food/files/plant/docs/pesticides_mrl_guidelines_wrkdoc_2019-12682.pdf (accessed 21 May, 2020).
Minimum criteria for chromatographic-mass spectrometric confirmation of the identity of analytes for doping control purposes (2015). Available at: https://www.wada-ama.org/sites/default/files/resources/files/td2015idcr_-_eng.pdf (accessed 21 May, 2020).
Acceptance criteria for confirmation of identity of chemical residues using exact mass data for the FDA foods and veterinary medicine program (2015). Available at: https://www.fda.gov/media/96499/download (accessed 21 May, 2020).
AORC guidelines for the minimum criteria for identification by chromatography and mass spectrometry (2016). Available at: http://www.aorc-online.org/documents/aorc-ms-criteria-modified-23-aug-16. (accessed 21 May, 2020).
Guidance for industry: Mass spectrometry for confirmation of the identity of animal drug residues (2003). Available at: https://www.fda.gov/media/70154/download. (accessed 21 May, 2020).
Milman B.L., Zhurkovich I.K. Identification performance of low-molecular compounds by searching tandem mass spectral libraries with simple peak matching. Mass Spectrom. Lett., 2018, vol. 9, no. 3, pp. 73-76. DOI: 10.5478/MSL.2018.9.3.73.
NIST 17 GC method / Retention index library. Available at: https://chemdata.nist.gov/dokuwiki/doku.php?id=chemdata:ridatabase (accessed 21 May, 2020).
CAS content. Available at: https://www.cas.org/about/cas-content (accessed 21 May, 2020).
PubChem. Available at: https://pubchem.ncbi.nlm.nih.gov (accessed 21 May, 2020).
ChemSpider. Available at: http://www.chemspider.com (accessed 21 May, 2020).
ZINC 15. Available at: http://zinc15.docking.org (accessed 21 May, 2020).
Milman B.L., Ostrovidova E.V., Zhurkovich I.K. Bol'shie himicheskie bazy dannyh svobodnogo dostupa v netselevom mass-spektrometricheskom analize [Big chemical databases in a modern non-target mass spectrometry analysis]. Mass-spektrometriia [Mass Spectrometry], 2020, vol. 17, no. 2, pp. 125-132 (In Russian).
Wiley registry of mass spectral data. Available at: https://www.sisweb.com/software/wiley-registry.htm (accessed 21 May, 2020).
The NIST 20 Mass spectral library. Available at: https://www.sisweb.com/software/ms/nist.htm (accessed 21 May, 2020).
Montenegro-Burke J.R., Guijas C., Siuzdak G. METLIN: a tandem mass spectral library of standards. Computational Methods and Data Analysis for Metabolomics. New York, Humana, 2020, pp. 149-163.
METLIN. Available at: https://metlin.scripps.edu/ms_ms_spectrum_match_search.php (accessed 21 May, 2020).
GNPS: Global natural products social molecular networking. Available at: https://dorresteinlab.ucsd.edu/gnps (accessed 21 May, 2020).
MoNA - MassBank of North America. Available at: https://mona.fiehnlab.ucdavis.edu (accessed 21 May, 2020).
MassBank. Available at: http://www.massbank.jp (accessed 21 May, 2020).
DOI: https://doi.org/10.15826/analitika.2020.24.3.003
Ссылки
- На текущий момент ссылки отсутствуют.
